 El consumo de sustancias tóxicas, entre ellas el alcohol, está frecuentemente presente en algunos delitos, siendo en algunas ocasiones el estado de ebriedad un atenuante e incluso un eximente de culpa (C.P arts 20 y 21), mientras que en otras ocasiones, puede representar per se un delito, como es el caso de los delitos contra la seguridad del tráfico (C.P arts 379-381).
El consumo de sustancias tóxicas, entre ellas el alcohol, está frecuentemente presente en algunos delitos, siendo en algunas ocasiones el estado de ebriedad un atenuante e incluso un eximente de culpa (C.P arts 20 y 21), mientras que en otras ocasiones, puede representar per se un delito, como es el caso de los delitos contra la seguridad del tráfico (C.P arts 379-381).
En el consumo de alcohol influyen múltiples factores ambientales (factores socioculturales y psicológicos, educación, enfermedades mentales, etc), y también genéticos. Así pues, se considera el abuso de alcohol como una enfermedad multifactorial, en la que están implicados genes y la interacción de éstos con el ambiente. Apenas se conocen los factores genéticos que pueden modular el consumo de alcohol, si bien estudios llevados a cabo con individuos adoptados y con gemelos han puesto de manifiesto que alrededor del 40-60% de la variación en la vulnerabilidad al alcoholismo tendría una base genética [1,2].
Actualmente se está investigando la implicación de diversos polimorfismos genéticos en genes candidatos relacionados con el metabolismo del etanol con la posible susceptibilidad genética al consumo de alcohol.
Autor
F. Francès1, JV. Sorlí2, A. Castelló1, F. Verdú3, D. Corella3 y O. Portolés4
1Profesor Ayudante. Facultad de Medicina. Universidad de Valencia.
2Profesor Asociado. Facultad de Medicina. Universidad de Valencia.
3Profesor Titular. Facultad de Medicina. Universidad de Valencia.
4Investigador Contratado. Dep. Llenguatges i Sistemes Informàtics. Universitat Jaume I (Castelló)
Dirección para correspondencia:
Francesc Francès Bozal
Dep. Medicina Preventiva i Salut Pública,
Ciències de l’Alimentació,Toxicologia i Medicina Legal.
Av. Blasco Ibáñez nº 15.
46010 València (España).
Tfno: 96 398 31 07
E-Mail: Esta dirección de correo electrónico está siendo protegida contra los robots de spam. Necesita tener JavaScript habilitado para poder verlo.
El alcohol es degradado por oxidación hepática, de forma predominante mediante el sistema alcohol deshidrogenasa (ADH) citosólica, que genera acetaldehído como resultado de su acción enzimática, el cual será substrato de la aldehído deshidrogenasa (ALDH) que lo degradará a su vez a acetato. Menos de un 10% del etanol sigue otras vías degradativas como la del sistema microsomal hepático y la catalasa. El primer paso del metabolismo del etanol en el organismo, podría explicar parte de la mayor o menor tolerancia al alcohol y señalar diferencias en el comportamiento del individuo frente a estas bebidas, debido a su mayor o menor toxicidad en función de su constitución genética respecto a este metabolismo. La ADH Clase I comprende tres enzimas: ADH1A, ADH1B y ADH1C, siendo la alcohol deshidrogenasa 1C (ADH1C, previamente llamada ADH3) la menos estudiada. La alcohol deshidrogenasa 1C, presenta dos isoformas, la gamma 1 y la gamma 2. Gamma 1 se caracteriza por presentar isoleucina (Ile) en el codón 349 y arginina (Arg) en el codón 279, frente a las formas gamma 2 que presentan valina (Val) y glutamina (Gln), respectivamente. [3]. Las formas gamma 1 codifican una enzima que presenta una velocidad de metabolización del alcohol dos veces y media mayor que las formas gamma 2, lo que implicaría una acumulación de su catabolito, el acetaldehido, [4,5] que es capaz de generar un cuadro metabólico conocido como “flushing” caracterizado por enrojecimiento facial, taquicardia y somnolencia [6] y que daría lugar a una respuesta de aversión al alcohol disminuyendo así el riesgo de consumo del mismo [7]. Por el contrario, aquellos individuos cuya velocidad de metabolización fuera menor (formas gamma 2) tendrían mayor riesgo de consumir alcohol al no sufrir “flushing” tras una ingesta alcohólica y prolongarse en el tiempo la presencia de etanol en sangre.
Si esta variante genética, junto a otras en otros genes como ADH2 y ALDH2, llegara a alcanzar el suficiente nivel de evidencia científica, quizá podría llevar consigo un replanteamiento de la voluntariedad en la ingestión de bebidas alcohólicas. En los casos en los que la ebriedad sea parte sustancial de un injusto típico, mediado la correspondiente prueba pericial, podría ser valorada por el tribunal correspondiente como circunstancia modificadora de la responsabilidad. De igual forma, también podría llegar a tener cierto valor procesal en los casos de reincidencia y en los casos de fracaso o abandono de tratamientos rehabilitadores.
El objetivo del presente trabajo ha sido estudiar la prevalencia de la variante genética gamma 2 (metabolizadores lentos), medida a través del polimorfismo Ile349Val en el gen de la ADH1C en población general mediterránea española, estudiar su asociación con el consumo de alcohol y reflexionar sobre el posible alcance de la implicación de estas variantes en el ámbito de la medicina legal.
Material y métodos
Individuos
Se ha llevado a cabo un estudio transversal en 869 individuos adultos residentes en la provincia de Valencia, con edades comprendidas entre 18 y 65 años. La muestra fue seleccionada aleatoriamente entre pacientes y acompañantes que acudieron a una consulta de un centro de Atención Primaria de Salud entre Febrero de 2000 y Mayo de 2001. Tras informar del propósito de nuestro estudio, se les solicitó su consentimiento [8]. Se obtuvieron muestras de sangre venosa periférica para realizar la extracción de ADN y se les administró un cuestionario para valorar el consumo de alcohol. En dicho cuestionario se preguntaba sobre la frecuencia de consumo semanal y de fin de semana de un listado de bebidas alcohólicas incluyendo cerveza, vino blanco, vino tinto y rosado, cava, carajillo, coñac, whisky, cubalibre, cazalla, martini, anís y vermut. [9]. Para el cálculo del consumo total de alcohol en gramos se utilizó la tabla de equivalencias de graduación alcohólica [10],
Genotipado de la variante Ile349Val
El ADN fue extraído mediante el método del fenol-cloroformo y posterior precipitación con etanol [11]. Para la detección de la variante genética denominada gamma 2, se determinó el polimorfismo Ile349Val ya que se encuentra en completo desequilibrio de ligamiento con la otra variante (Arg279Gln), en el mismo gen. Para ello se realizó un reacción en cadena de la polimerasa (PCR) en un volumen total de 25 μl , con 30 ng de ADN, 2 mM de MgCl2, 10 mM de Tris HCl (pH=9), 0,1 μM de cada cebador (5’-GCT TTA AGA GTA AAT ATT CTG TCC CC-3’ y 5’-AAT CTA CCT CTT TCC GAA GC-3’) de acuerdo con el diseño de Groppi et al [12], 12 μM de cada desoxinucleótido trifosfato y 0,5 unidades de Taq polimerasa (Promega Madison, USA). El producto de PCR fue digerido con el enzima de restricción SspI (Promega, Madison, USA) a 37ºC durante 4 horas. Los productos digeridos fueron sometidos a electroforesis en un gel de agarosa al 3% y visualizados mediante luz ultravioleta tras tinción con bromuro de etidio. Los diferentes genotipos se identificaron en función de la presencia o ausencia de sitio de restricción para SspI. Así, la presencia del alelo Ile generó un sitio de restricción SspI dando lugar a dos fragmentos de 67 y 63 pares de bases (pb) mientras que el alelo 349Val se caracterizó por la ausencia de sitio de restricción para SspI y por tanto por la presencia de un sólo fragmento no digerido de 130 pb.
Análisis estadístico
Para la comparación de medias se utilizaron el test T de Student y el test de ANOVA según se compararan medias de dos o más grupos para comparar variables continuas y el test de χ2 para comparar variables categóricas. La prueba χ2 se utilizó también para comparar las frecuencias observadas y esperadas asumiendo el equilibrio de Hardy-Weinberg.
Se definió como consumidor habitual de alcohol aquellos individuos que consumían bebidas alcohólicas de manera regular al menos una vez a la semana. Para estudiar la asociación con el consumo de alcohol se consideraron modelos dominantes, codominantes y recesivos, y de acuerdo con ello se agruparon los portadores de las variantes de interés. Para estimar el riesgo de consumo de alcohol según dicho polimorfismo se utilizaron modelos de regresión logística, calculando la odds ratio (OR) y su intervalo de confianza (IC) al 95%. Se llevaron a cabo modelos de regresión crudos y ajustados por posibles factores de confusión.
Se calculó el nivel de significación de p bilateral para cada test y se consideró estadísticamente significativo un valor de p<0,05.
Resultados
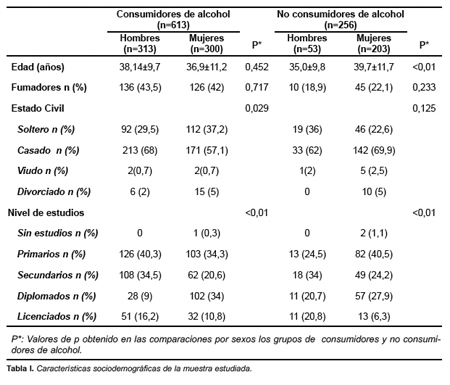
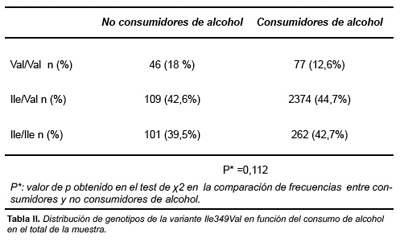
Las características sociodemográficas de la muestra estudiada se indican en la tabla 1. El 85,5% de los hombres y el 59,6% de las mujeres fueron clasificados como consumidores habituales de alcohol (p<0,01). La prevalencia de genotipos en la población estudiada fue de 360 (41%) Ile/Ile, 387 (44,5%) Ile/Val y 122 (14%) Val/Val. La distribución de genotipos no se mostró discrepante con la distribución de Hardy-Weinberg (p= 0,21). No existieron diferencias por género en el genotipo ADH1C. No se encontraron diferencias de frecuencias en la distribución genotípica de las variantes de ADH1C entre bebedores y no bebedores (tabla 2).
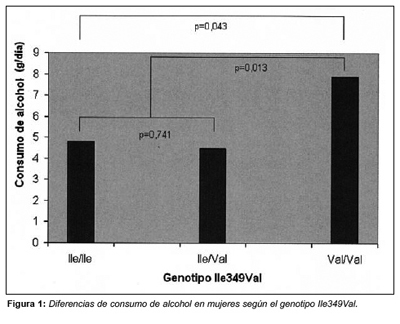
Sin embargo, entre los bebedores, al estudiar la cantidad de alcohol consumido (media en g/día), sí que se observaron asociaciones estadísticamente significativas entre dicho consumo y los genotipos de la ADH1C, fundamentalmente en mujeres.
Así, al considerar sólo mujeres bebedoras habituales de alcohol, se obtuvo que la media diaria de dicho consumo fue de 7,9±11,7 g/día en las metabolizadoras lentas (Val/Val) frente a 4,5±5,2 g/d en las Ile/Val y 4,8 g/d±6,3 g/día en las Ile/Ile (p=0,043). Esta asociación sugiere un patrón recesivo de asociación en el que es necesario poseer los dos alelos mutados para que se manifiesten los efectos. Al agrupar las homozigotas normales y las portadoras en heterocigois de la variante Val y comparar los efectos se obtuvo los siguientes valores de consumo de alcohol: 4,6±5,7 g/día en portadores Ile frente a 7,9±11,7 g/día en los Val/Val (p=0,013) (Fig. 1)
Al analizar los tipos de bebidas alcohólicas según este patrón recesivo, destacan las grandes diferencias obtenidas en las frecuencias de consumo de vino tinto entre semana, que fue tres veces más consumido en mujeres Val/Val, en comparación con las portadoras Ile (p=0,001). Cabe destacar las grandes diferencias en el consumo de cubalibres (de ginebra o vodka), tanto entre semana (p= 0,030) como en el fin de semana (p= 0,001).
En los hombres no se observaron diferencias estadísticamente significativas en las medias de consumo de alcohol entre los bebedores, en función del polimorfismo, que fueron de 13,4±12,2 g/día en Val/Val, 12,9±13,4 g/día en Ile/Val y 15,2±13,7 g/día en Ile/Ile (p=0,384), aunque para alguna de las bebidas analizadas como el whisky entre semana y la caña de cerveza en fin de semana, sí que se observaron mayores frecuencias de consumo en los Val/Val, ambos en el límite de la significación estadística (p=0,054).
Finalmente se estimó el riesgo de consumo elevado de alcohol (entendido éste como el doble de la media de consumo de la población femenina: 2X5 g/día=10 g/día) entre las mujeres bebedoras asociado a la variante Val/Val. Se observó (tabla 3) que la prevalencia del genotipo Val/Val fue del 20,5% en mujeres bebedoras de más de 10 g/día, frente a 9,3% en mujeres bebedoras de menos de 10 g/día (p=0,038). El mayor riesgo de consumo elevado de alcohol, más de 10 g/día, asociado al genotipo Val/Val en mujeres (OR= 2,51 p= 0,044) permaneció estadísticamente significativo tras ajustar por consumo de tabaco, edad, estado civil y nivel de estudio (OR= 2,59; p= 0,048). En cambio en los hombres, la magnitud de la asociación fue mucho menor, también ajustada por las mismas variables, y no alcanzó la significación estadística (OR=1,15; p= 0,711).

Discusión
En nuestro estudio hemos valorado el posible efecto de un polimorfismo situado en el gen de la alcohol deshidrogenasa 1C y el riesgo de consumo de alcohol. La frecuencia del alelo gamma 2 en nuestra población se encuentra ligeramente por debajo de las descritas en otras poblaciones europeas [13].
Aunque dicho polimorfismo no presentaba diferencias entre bebedores y no bebedores, al considerar la cantidad de alcohol consumida por los bebedores, sí que encontramos asociación de este polimorfismo con un mayor consumo de alcohol.
Según nuestros resultados, las mujeres consumidoras habituales de alcohol que eran homozigotas para el alelo gamma 2 (Val/Val) prácticamente duplicaron el promedio de consumo de alcohol frente a las portadoras de la variante gamma 1 (Ile). Además, las homozigotas gamma 2 presentaban una mayor frecuencia de consumo de determinadas bebidas alcohólicas. Concretamente triplicaron el consumo de vino tinto entre semana presentando también frecuencias significativamente mayores de consumo de cubalibre tanto entre semana como en los fines de semana. Todos estos hechos se traducen en un riesgo 2 veces y medio superior en estas mujeres homozigotas para la variante gamma 2, de consumir alcohol por encima de 10 g/día. Estos resultados están en concordancia con el trabajo de Ebrahim et al [14] realizado en la población femenina participante en el estudio Brithish Women’s Heart and Health (BWHHS), estos autores hallaron que las homozigotas gamma 2 (Val/Val), presentaban un mayor consumo de bebidas alcohólicas que las portadoras gamma 1 (Ile) con diferencias estadísticamente significativas (p= 0,03).
En los hombres no se observaron diferencias estadísticamente significativas de consumo promedio de alcohol según genotipo, aunque los homozigotos (Val/Val) sí consumieron algunas bebidas alcohólicas con mayor frecuencia, con diferencias cercanas a la significación estadística.
Estos resultados se muestran concordantes con la hipótesis de la velocidad de metabolización del alcohol. Esta hipótesis, verificada con bastante consistencia para el caso de otra enzima denominada ADH1B [15,16], sugiere que las formas metabolizadoras rápidas (ADH1B*2 y *3), serían protectoras frente al riesgo de alcoholismo. Ahora bien, en el caso de ADH1C, aunque la evidencia no es tan constante [17,18,19], la mayoría de estudios sugiere que la isoforma metabolizadora lenta de la ADH1C o gamma 2 estaría asociada a mayores consumos de alcohol [20-23] ya que las bajas concentraciones de acetaldehído generadas no provocarían manifestaciones tóxicas y por tanto no existiría estímulo de aversión al alcohol. Posiblemente la explicación de esta menor consistencia en los resultados comparando ADH1B y ADH1C, radique en la magnitud de la diferencia de velocidad metabolizadora entre las diversas isoformas, siendo de 100 veces en ADH1B, mientras que en ADH1C únicamente es de 2,5 veces. Esto podría explicar también el menor impacto de la variante ADH1C en hombres que en mujeres, ya que en nuestro estudio la asociación más relevante se ha encontrado sólo en mujeres.
Nuestros resultados, junto con otros trabajos anteriores, ofrecen datos que podrían tenerse en cuenta en la valoración de la importancia del sustrato genético en la predisposición al consumo de este tóxico. Así pues, siendo la intoxicación etílica motivo de infracción penal en determinados casos, exista o no perjuicio a terceros, en un futuro podría plantearse la oportunidad del estudio del perfil de genes asociados al consumo de alcohol ante personas imputadas por realizar determinadas actividades en estado de ebriedad, reflexionando sobre la posibilidad de tenerlo en cuenta a la hora de enjuiciar el caso, para así matizar la voluntariedad e imputabilidad de dicho acto ilícito. En previsión de este hecho, consideramos necesarios más estudios epidemiológicos sobre la influencia de otras variantes genéticas en el consumo de alcohol, para conocer mejor la posible susceptibilidad genética. Mientras éstos no se lleven a cabo y se alcance el suficiente nivel de evidencia, estas características no deberían ser admitidas como prueba en procedimientios judiciales. q
Bibliografía
1.- Heath AC, Bucholz KK, Madden PA, Dinwiddie SH, Slutske WS, Bierut LJ, Statham DJ, Dunne MP, Whitfield JB, Martin NG. Genetic and environmental contributions to alcohol dependence risk in a national twin sample: consistency of findings in women and men. Psychol Med 1997; 27:1381-96.
2.- Kendler KS, Heath AC, Neale MC, Kessler RC, Eaves LJ. A population-based twin study of alcoholism in women. JAMA 1992; 14; 268:1877-82.
3.-Xu YL, Carr LG, Bosron WF, Li TK, Edenberg HJ. Genotyping of human alcohol dehydrogenases at the ADH2 and ADH3 loci following DNA sequence amplification. Genomics 1988; 2:209–214.
4.- Thomasson HR, Crabb DW, Edenberg HJ, Li TK. Alcohol and aldehyde dehydrogenase polymorphisms and alcoholism. Behav Genet 1993; 23:131-6.
5.- Wall TL, Garcia-Andrade C, Thomasson HR, Cole M, Ehlers CL. Alcohol elimination in Native American Mission Indians: an investigation of interindividual variation. Alcohol Clin Exp Res 1996; 20:1159-64.
6.- Harada S, Agarwal DP, Goedde HW, Tagaki S, Ishikawa B. Possible protective role against alcoholism for aldehyde dehydrogenase isozyme deficiency in Japan. Lancet 1982; 9; 2:827.
7.- Takeshita T, Morimoto K, Mao X, Hashimoto T, Furuyama J. Characterization of the three genotypes of low Km aldehyde dehydrogenase in a Japanese population. Hum Genet 1994; 94:217-23.
8.- Sorli JV, Velert R, Guillen M, Portoles O, Ramirez JB, Iborra J, Corella D. Efecto del polimorfismo de la Apolipoproteína E en el perfil lipoproteico y riesgo cardiovascular en una población mediterránea. Med Clin 2002; 118: 569-74.
9.- Corella D, Saiz C, Guillen M, Portoles O, Mulet F, Gonzalez JI, Ordovas JM. Association of TaqIB polymorphism in the cholesteryl ester transfer protein gene with plasma lipid levels in a healthy Spanish population. Atherosclerosis. 2000;152: 367-76.
10.- Cuevas Badenes J, Sanchís Fortea M. Tratado de alcohología. NILO Industria Gráfica SA, 2000
11.- Sambrook J, Fritsch EF; Maniatis T. Molecular Cloning, a laboratory manual. Second Edition; 1989. Cold Spring Harbor Lab. Press.
12.- Groppi A, Begueret J, Iron A. Improved methods for genotype determination of human alcohol dehydrogenase (ADH) at ADH2 and ADH3 loci using polymerase chain reaction-directed mutagenesis. Clin Chem 1990; 36:1765–8.
13.- Borras E, Coutelle C, Rosell A, Fernandez-Muixi F, Broch M, Crosas B, Hjelmqvist L, Lorenzo A, Gutierrez C, Santos M, Szczepanek M, Heilig M, Quattrocchi P, Farres J, Vidal F, Richart C, Mach T, Bogdal J, Jornvall H, Seitz HK, Couzigou P, Pares X. Genetic polymorphism of alcohol dehydrogenase in europeans: the ADH2*2 allele decreases the risk for alcoholism and is associated with ADH3*1. Hepatology 2000; 31: 984-9.
14. Ebrahim S, Lawlor DA, Shlomo YB, Timpson N, Harbord R, Christensen M, Baban J, Kiessling M, Day I, Gaunt T, Davey Smith G. Alcohol dehydrogenase type 1C (ADH1C) variants, alcohol consumption traits, HDL-cholesterol and risk of coronary heart disease in women and men: British Women's Heart and Health Study and Caerphilly cohorts. Atherosclerosis 2007 20; [Epub ahead of print]
15.- Ehlers CL, Montane-Jaime K, Moore S, Shafe S, Joseph R, Carr LG. Association of the ADHIB*3 allele with alcohol-related phenotypes in Trinidad. Alcohol Clin Exp Res 2007; 31:216-20.
16.- Lorenzo A, Auguet T, Vidal F, Broch M, Olona M, Gutierrez C, Lopez-Dupla M, Sirvent JJ, Quer JC, Santos M, Richart C. Polymorphisms of alcohol-metabolizing enzymes and the risk for alcoholism and alcoholic liver disease in Caucasian Spanish women.Drug Alcohol Depend 2006; 15;84:195-200.
17.- Choi IG, Son HG, Yang BH, Kim SH, Lee JS, Chai YG, Son BK, Kee BS, Park BL, Kim LH, Choi YH, Shin HD. Scanning of genetic effects of alcohol metabolism gene (ADH1B and ADH1C) polymorphisms on the risk of alcoholism. Hum Mutat 2005; 26:224-34.
18.- Wall TL, Carr LG, Ehlers CL. Protective association of genetic variation in alcohol dehydrogenase with alcohol dependence in Native American Mission Indians. Am J Psychiatry 2003; 160: 41-6.
19.-Saccone NL, Kwon JM, Corbett J, Goate A, Rochberg N, Edenberg HJ, Foroud T, Li T-K, Begleiter H, Reich T, Rice JP: A genome screen of maximum number of drinks as an alcoholism phenotype. Am J Med Genet 2000; 96:632-637.
20.-Matsuo K, Hiraki A, Hirose K, Ito H, Suzuki T, Wakai K, Tajima K. Impact of the Alcohol-Dehydrogenase (ADH) 1C and ADH1B polymorphisms on drinking behavior in nonalcoholic Japanese. Hum Mutat 2007;28:506-510.
21.- Montane-Jaime K, Moore S, Shafe S, Joseph R, Crooks H, Carr L, Ehlers CL. ADH1C*2 allele is associated with alcohol dependence and elevated liver enzymes in Trinidad and Tobago. Alcohol 2006; 39:81-6.
22.- Konishi T, Luo HR, Calvillo M, Mayo MS, Lin KM, Wan YJ. ADH1B*1, ADH1C*2, DRD2 (-141C Ins), and 5-HTTLPR are associated with alcoholism in Mexican American men living in Los Angeles. Alcohol Clin Exp Res 2004; 28(8):1145-52.
23.- Konishi T, Calvillo M, Leng AS, Feng J, Lee T, Lee H, Smith JL, Sial SH, Berman N, French S, Eysselein V, Lin KM, Wan YJ. The ADH3*2 and CYP2E1 c2 alleles increase the risk of alcoholism in Mexican American men. Exp Mol Pathol 2003; 74:183-9.
Fuente: Francès F., Sorlí JV., Castelló A., Verdú F., Corella D., Portolés O.. Predisposición genética en el consumo de alcohol: el caso de la Alcohol Deshidrogenasa 1C. Cuad. med. forense. [periódico en la Internet]. 2007 Jul [citado 2008 Sep 14] ; (48-49): 157-164. Disponible en: http://scielo.isciii.es/scielo.php?script=sci_arttext&pid=S1135-76062007000200004&lng=es&nrm=iso.

